共同通讯联系人:郗冬梅,教授; Tel/Fax:0539-8766862; E-mail:dongmxi@126.com; 研究方向:单分子分析
生物纳米孔传感技术因其快速、低成本、无需荧光标记等优点,在化学和生物等诸多研究领域得到广泛应用,已发展成为一种新颖的、独具特色的单分子分析手段。 该技术目前主要应用于DNA测序研究,同时在单分子分析领域也取得了令人瞩目的成就。 该文简要介绍了生物纳米孔分析技术的原理和生物孔的种类,主要总结了近20年来生物纳米孔在DNA测序和单分子分析中的研究进展并予以了展望。
Co-corresponding author:XI Dongmei, professor; Tel/Fax:0539-8766862; E-mail:dongmxi@126.com; Research interests:single-molecule analysis
Biological nanopore sensing has been developed as an attractive single-molecule tool with advantages of being rapid, low-cost, and label-free. Nanopores are not only being widely developed for DNA sequencing, but they also have been utilized to analyze a wide range of analytes at the single-molecule level. This review summarizes and prospects the principle of the nanopore analysis and nanpore category, with an emphasis on the progress in nanopore-based DNA sequencing and single-molecule analysis.
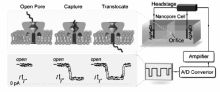
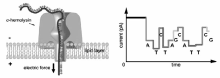
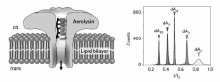
纳米孔分析技术是从20世纪90年代中期开始发展起来的新型单分子分析手段。 1996年,Deamer、Branton和Kasianowicz等首次报道了利用金黄色葡萄球菌 α-溶血素( α-Hemolysin, α-HL)天然生物通道蛋白获得单个单链DNA(ssDNA)分子和RNA分子的阻断电流信号[1]。 这项具有划时代意义的研究为纳米孔分析技术奠定了基础,之后的20年,该技术凭借其快速、低成本、无需荧光标记等优势,在化学和生物等诸多研究领域得以广泛应用[2,3]。 工作原理如图1所示,检测池内加入高盐溶液,在纳米孔两端施加一个外加电场后,溶液中的阴/阳离子发生定向移动,产生恒定的电流。 由于生物纳米孔的直径多为1.0~3.0 nm,因此在电场力驱动下只能容许单个待测物分子进入并穿过纳米孔,造成电流的阻断。 不同待测物分子产生阻断电流的时间、程度、频率及形状不同。 通过对大量阻断电流信号的统计,可在单分子水平获取分子的结构、组成、尺寸及电荷等信息[4]。
根据构建纳米孔的蛋白质不同,生物纳米孔主要分为 α-HL[1]、气单胞菌溶素(Aerolysin)[5]、耻垢分枝杆菌毒素蛋白(Mycobacterium smegmatisporin A,MspA)[6]和噬菌体phi29连接器(Bacteriophage phi29 connector)[7]等几类(图2)。 下面分别做简要介绍。
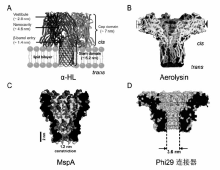 | 图2 纳米孔结构示意图[6,7,8,9]Fig.2 The structure of nanopores[6,7,8,9]A. α-HL[8]; B.Aerolysin[9]; C.MspA[6]; D.Phi29connector[7] |
α-HL生物通道蛋白被用于构建了第一个纳米孔[1],也是目前应用最为广泛的生物纳米孔。 α-HL是金黄色葡萄球菌分泌的一种多肽毒素,含有293个氨基酸,相对分子质量为3.32×104[10],可通过自组装形成七聚体的跨膜通道(图2A),这一蘑菇状的纳米通道由一个较大的膜外前庭和一个 β-桶状结构的跨膜茎部组成[10], cis端前庭入口处直径为~2.6 nm,前庭内腔直径为~4.6 nm,最窄处直径为~1.4 nm,trans端入口处直径为~2.0 nm。 而生物大分子的一级结构尺寸或大于该纳米孔最窄处直径、或与其相当,以核酸为例,poly C在中性pH条件下形成的单链直径为1.3 nm[11],柔性多肽链的直径为8.0~12 nm[12]。 因此, α-HL纳米孔的最窄处限制了多个待测生物大分子的进入,即一次只能通过一个生物大分子,确保了其能够进行单个分子的分析研究。 α-HL优异的性质使其成为目前应用最为广泛的生物纳米通道:第一, α-HL易于自组装且结构稳定,具有均一的电导,从而使得待测物产生的阻断事件具有可重复性;第二, α-HL对待测物基本不具有选择性,确保电流阻断事件是由单个待测分子的体积效应造成的;第三, α-HL内腔易于进行基因突变或化学修饰,实现特异性检测[13,14]。
Aerolysin是从嗜水气单胞菌中提取的 β成孔蛋白,是一种与人体腹泻病症以及深度伤口相关的革兰氏阴性杆菌属,成熟的Aerolysin可与真核细胞膜表面的糖蛋白受体相结合,并插入细胞膜形成一个孔道,改变细胞膜的通透性并最终导致细胞死亡[15]。 在水溶液中Aerolysin单体会自发地形成一个具有蘑菇状外形的七聚体结构(图2B),宽度为~15 nm,高度为~7.0 nm,形成的孔道直径为1.0~1.7 nm,其形状与目前在纳米孔技术中广泛应用的 α-HL类似,但在 cis端没有前庭结构[16]。 Aerolysin最早于2006年首次应用于纳米孔生物传感器,进行 α-螺旋胶原蛋白结构的研究[5],随后被用于分析蛋白的折叠和去折叠动力学过程[12]、多肽[17]、蛋白结构[18]、低聚糖的聚集程度和酶降解的动力学[19]等。
1996年,Kasianowicz等[1]证明了ssDNA和RNA分子能通过 α-HL蛋白质组成的孔道,并且能检测到它们通过这种纳米级孔道时所造成的电流改变。 基于此现象,研究人员提出根据不同种类碱基通过纳米孔时产生的特征弱离子流信号,即可读出每条单链DNA上的序列信息(图3),并最终实现单分子测序。 这种基于纳米孔的DNA测序方法因快速、无需扩增、无需荧光标记就可直接获取碱基序列信息而被视为最具前景的“第三代DNA测序技术(Third-Generation DNA Sequencing)”,有望实现仅花费1000美金即可在24 h内测定个人基因组的完整序列[2]。 然而,真正将该技术应用于DNA测序,还面临着巨大的挑战。
1)ssDNA通过纳米孔的速度极快,每个碱基约1.0~10 μs[1,11],造成的阻断电流信号极小,仅有皮安级,阻碍了纳米孔测序技术的发展。 要达到在皮安级电流水平上检测单个核苷酸的精度,必须延缓DNA通过纳米孔的速度。 目前,人们已经尝试了许多实验策略,如增加待测溶液的粘度[24]、降低待测溶液的温度[25]以及采用基因突变技术改变 α-HL纳米孔的氨基酸电荷分布[26]等,最有效的办法之一是利用工具酶作为分子马达控制DNA分子穿过纳米孔。 DNA聚合酶聚合每一个碱基的时间长达数十毫秒,可以像齿轮一样每次只驱动ssDNA链上的一个核苷酸运动[27]。 如图4所示,噬菌体phi29 DNA聚合酶以每秒2.5~40碱基的速度转动ssDNA进入 α-HL纳米孔[27,28],每一条DNA链能够自动在DNA聚合酶校对过程中进入纳米孔,并在DNA聚合过程中退出纳米孔,从而实现纳米孔对每一个碱基进行两次测量,提高了 α-HL纳米孔DNA测序的时间分辨率及准确性。
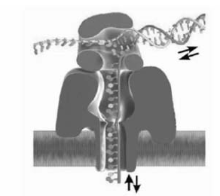 | 图4 Phi29 DNA聚合酶控制ssDNA穿过 α-HL纳米孔[27]Fig.4 Translocation speed of ssDNA through an α-HL is controlled by phi29 DNA polymerase[27] |
2)单个碱基阻断电流的分辨率低,需放大每一个脱氧核苷酸造成的阻断电流信号并增大4种脱氧核苷酸阻断电流的差异,从而提高纳米孔DNA测序的准确度。 研究表明, α-HL的 β-桶状结构中有3个位点决定了核苷酸阻断电流的程度[29],通过基因突变的方法能提高这3个位点对碱基的识别能力,从而增大阻断电流差异。 β-环糊精不仅能包裹单个碱基,且尺寸与 α-HL纳米孔的最窄处匹配,将环糊精嵌入基因修饰后的 α-HL纳米孔,可以有效缩短纳米孔最窄处的直径,提高对不同碱基的分辨率[30,31]。 如图5所示,Bayley等[32]利用外切酶I剪切ssDNA形成的单个脱氧核糖核苷酸在电场力作用下进入 β-环糊精修饰的 α-HL纳米孔,能够实现高达99.8%的单碱基分辨率。 此外,Kumar等[33]提出基于纳米孔的“边合成边测序(nanopore-based sequencing by synthesis)”策略,用不同长度的聚乙二醇(PEG)分别标记4种不同的碱基,检测与模板链配对后释放的标记物,以此实现对单个碱基的超灵敏分辨。
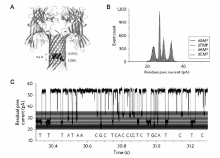 | 图5 环糊精修饰 α-HL纳米孔进行DNA测序方法示意图[32]Fig.5 The cyclodextrin modified α-HL nanopore for DNA sequencing[32] A.the structure of α-HL modified by cyclodextrin; B.residual current histogram of nucleotide; C.discrimination of the nucleotides based on the residual current |
由于 α-HL纳米孔易于修饰且具有可重复的空间结构,迄今为止大部分生物纳米孔DNA测序研究均是基于 α-HL纳米孔进行的。 然而, α-HL纳米孔的茎部长度~5.2 nm,可容纳~20个核苷酸。 这种结构使得ssDNA分子通过 α-HL纳米孔时产生的阻断电流信号是~20个核苷酸的整体阻断效应造成的,而非单个核苷酸的体积效应引起的,难以从阻断电流信息中清晰地得出每一个碱基的阻断电流信息。 针对这一问题,通道蛋白MspA被应用于DNA测序研究中,被认为是目前在DNA测序方面最有前景的生物纳米孔[6,20]。 Manrao等[34]将MspA纳米孔与phi29 DNA聚合酶DNA测序方法相结合,可控制ssDNA以每个碱基~28 ms的速度进入MspA纳米孔并产生极易分辨的阻断电流信号,4种脱氧核苷酸之间的阻断电流差异高达40 pA(图6)。
 | 图6 MspA纳米孔与phi29 DNA聚合酶结合读取DNA序列[34,35]Fig.6 Reading DNA with MspA nanopore and phi29 DNA polymerase[34,35] A.diagram of the combination between MspA nanopore and phi29 DNA polymerase; B.reading DNA with the nanopore |
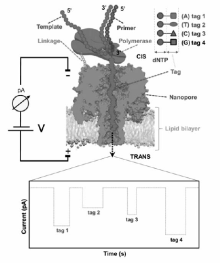
最近,George Church教授和他的团队[36]开发出了一种新的基于生物工程纳米孔的DNA测序电子平台(图7),这一测序工具包含磷脂膜上由6个亚基构成的开孔孔道,还有一个特定的亚基上结合了DNA聚合酶(Polymerase),可以合成一串与待测模版链互补的DNA链。 通过扩增预先连接在模版链上的引物(Primer),聚合酶添加互补的核苷酸(dNTP),每一种核苷酸都携带了特定的标记物(Tag)。 当聚合酶按照模板逐步合成时,标记物先后释放到纳米孔中引起特征电流的变化(V)。
寡聚核苷酸链会在 α-HL纳米孔的前庭经历由弱相互作用变化引起的解链或者去折叠的过程,降低其构象熵垒后,以伸展的形式通过纳米孔最窄处。 单个ssDNA或RNA分子在 α-HL纳米孔内经历解链或去折叠过程时,能够产生特征性阻断电流信号。 通过分析特征信号,可在实现核酸分子构象的单分子水平研究。 1999年,Akeson等[11]利用 α-HL纳米孔检测了单个RNA分子由构象引起的阻断事件差异,由于polyA具较强的碱基堆积作用,可形成 α螺旋结构,因此其产生的阻断时间大于polyC和polyU。 同时,Kasianowicz小组[37]的研究表明,poly(dT)较弱的碱基堆积作用使其穿过 α-HL纳米孔时,产生二阶特征阻断事件,而poly(dC)和poly(dA)由于具有刚性的二级结构,只产生脉冲状阻断事件“pulse-like signals”。
除了用于核酸的构象研究,生物纳米孔在DNA和RNA检测方面也表现出极大的优势。含有前导寡聚核苷酸链的dsDNA在电压驱动下,前导链率先进入 α-HL纳米孔前庭,随后伸入 β-桶状结构,同时引导dsDNA进入 α-HL纳米孔前庭并最终导致自发解链过程,以ssDNA形式穿过 α-HL纳米通道[38,39]。 研究结果表明,由于杂交双链的稳定性差异,具有前导寡聚核苷酸链且含有单碱基错配的dsDNA穿过纳米孔需要的时间小于完全互补配对的dsDNA。 据此,可以利用 α-HL纳米孔检测单个dsDNA分子的单碱基错配。 Gu等[40]针对肺癌相关microRNA(miRNA)设计了带有前导链的寡核苷酸探针,二者的杂交产物穿过 α-HL纳米孔时能够产生特征性阻断信号,信号频率与靶miRNA的浓度呈正相关,可用于检测血液样品中的miRNA,并根据阻断时间的差异区分miRNA单碱基差异,首次实现了 α-HL纳米孔分析技术在实际样品检测中的应用(图8)。 我们课题组[41]将生物纳米孔与核酸类似物-锁核酸(locked nucleic acid,LNA)修饰的探针相结合(图9),当LNA探针与靶标let-7b的杂交产物经过 α-HL纳米孔时,会产生超长的阻断信号,而与含碱基错配的miRNA杂交、穿越纳米孔时,阻断时间非常短,因而从视觉上即可将单碱基错配明显区分,极大地提高了miRNA检测的特异性。 最近,Gu等[42]将双向电泳原理与纳米孔结合,利用聚阳离子纳米载体选择性结合靶DNA或RNA,而非目标分子在电压作用下反方向远离纳米孔,从而实现了核酸的抗干扰检测。 与常用的 α-HL纳米孔相比,Long等[43]首次证明Aerolysin纳米孔在寡聚核苷酸检测方面表现出更强的空间和时间分辨能力(图10),使用野生型Aerolysin生物孔可将ssDNA的过孔速度提高到每个碱基~2.0 ms,达到了对仅有单个核苷酸差异DNA分子的超灵敏识别,并实现了混合复杂体系的超灵敏检测和核酸外切酶Ι“步步降解”单链DNA过程的实时观测。
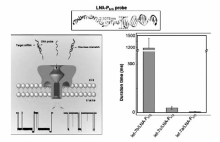 | 图9 基于 α-HL纳米孔利用LNA探针区分miRNA单碱基错配[41]Fig.9 LNA probe-enabled discrimination of miRNAs with single-nucleotide difference in α-HL nanopore[41] |
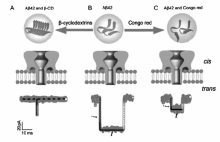
经过20多年的发展,基于纳米孔的DNA测序和核酸单分子检测已取得了巨大进展。 与此同时,利用该技术对蛋白质的分析也引起了很多研究者的关注。 与DNA相比,蛋白质分子的结构更加复杂,蛋白的电荷和疏水性相当多变,而且存在大量的二级和三级结构。 由于多肽电荷分布以及结构上的复杂性,使其进入 α-HL纳米孔的能量壁垒较高[44],通过在 α-HL的 β-桶状结构两侧进行氨基酸定点突变,改变 α-HL电荷分布,可调控 α-HL对多肽的捕获能力[45,46]。 Long等[47]利用 α-HL纳米孔在单分子水平实时观察阿兹海默症致病蛋白(A β42)结构变化,获得A β42单体及其寡聚物在纳米孔中的结构变化信息。 A β42单体自聚集形成的寡聚物能够被纳米孔捕获,产生较大的阻断电流信号;加入小分子药物刚果红调控后,可抑制A β42的聚集,形成结构较小的单体,单体穿过纳米孔产生的阻断电流值较小;而 β-环糊精能够促进A β42的快速聚集,形成较大体积聚集物,无法进入纳米孔(图11)。 此外,利用 α-HL纳米孔可实现在单分子水平上对帕金森症(Parkinson Disease,PD)致病蛋白 α-synuclein的纤维化行为进行分析[48]。 这为疾病的早期诊断和治疗提供一种全新的检测分析方法,并对蛋白与药物的相互作用机理等研究具有重要的指导意义。
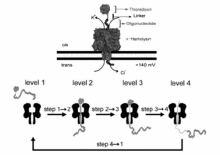
部分蛋白质在引导链的牵引下能够自发地解折叠并穿过纳米孔,有些蛋白质则需要更强的力使其变性并最终穿过纳米孔道。 目前,人们已经开始利用纳米孔检测蛋白的状态和特性。 Bayley等[49]以硫氧还蛋白为研究对象,其上连接寡核苷酸作为引导链牵引蛋白质解折叠并穿过纳米孔(图12),提出四步穿越机制。 2014年,Rosen等[50]证实 α-HL纳米孔能够区分硫氧还蛋白的不同磷酸化亚型,向单分子蛋白分析迈进了一步。 此外,Soskine等[51]利用基因工程手段构造了更大尺寸的生物纳米孔,允许折叠蛋白或结构域进入,以便分析蛋白与其他蛋白、药物或底物的相互作用。 对于纳米孔基础上的蛋白单分子测序而言,最关键的是设计马达一次拉动一个氨基酸穿过纳米孔。 现在纳米孔技术还很难做到将组成蛋白质的氨基酸进行一一解析,但是能够通过识别结构域等特征鉴定蛋白质,检测蛋白所处的状态或者评估蛋白之间的互作。 未来我们期待纳米孔技术能够早日成为单分子水平上研究蛋白质的成熟工具[52,53]。
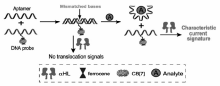
除了结构分析,近年来,生物纳米孔也开始用于蛋白质的定量检测。 由于蛋白质的尺寸大于生物孔的孔径,无法直接进行纳米孔分析,如何将其转换成DNA信号是解决问题的关键。 Li等[54]通过靶标结合诱导的链置换反应,利用 α-HL孔实现了对血小板衍生生长因子(PDGF-BB)的检测,检测限达500 fmol/L。 Wu等[55]设计了修饰二茂铁-葫芦[7]脲复合物(ferrocene ⊂ cucurbit[7]uril(Fc ⊂ CB[7]) )的DNA探针,待测物存在时与其适体特异性结合,导致适体-探针杂交物解链,释放的探针穿过 α-HL纳米孔时能够产生特征性的电流信号,从而实现了对血管内皮生长因子VEGF和凝血酶的高灵敏检测研究(图13)。 这一方法克服了纳米孔技术检测蛋白质等生物大分子的局限,进一步拓宽了纳米孔检测的底物种类。
用生物纳米孔直接检测有机小分子有一定的困难,如果能在纳米孔中引入分子适配器,利用适配器与待测物之间的特异性相互作用,可使有机小分子的检测成为可能。 适配器通常是环状分子,在纳米孔内部与孔壁以非共价键方式连接,成为纳米孔与小分子之间作用的媒介。 1999年,Bayley等[31]首次利用 α-HL纳米孔进行金刚烷分子的检测,在体系中加入与 α-HL孔径匹配的分子适配器— β-环糊精, β-环糊精可在纳米孔内停留长达数十毫秒,为监测金刚烷与环糊精的相互作用提供了足够的时间。 除 β-环糊精外,其它一些环状分子,如环状多肽、葫芦脲等也可用作适配器[56]。
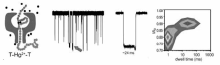
目前,将纳米孔技术用于金属离子检测的报道并不多见。 2000年,Braha等[57]利用基因工程手段改造 α-HL纳米孔,在孔内部引入4个组氨酸突变,其侧链上所带的咪唑基可与多种二价过渡金属离子如锌离子Zn(Ⅱ)、钴离子Co(Ⅱ)、镉离子Cd(Ⅱ)相互作用,由于不同金属离子与咪唑基的络合能力不同,因而进行纳米孔分析时能够分别产生相应的特征性阻断电流信号,从而首次实现了多种金属离子的同时定性分析和定量检测。 2011年,Wu等[58]设计了DNA分子作为探针,二价汞离子(Hg2+)存在时,富含胸腺嘧啶(T)的单链DNA由于T-Hg2+-T的键合作用能够形成含部分双链的发夹结构,该结构穿过 α-HL纳米孔时的阻断时间明显长于单链DNA的阻断时间(图14),利用该差别即可实现对Hg2+的快速检测和定量分析,检测限低至7.0 nmol/L。 采用类似的策略,铅离子(Pb2+)能够与富含鸟嘌呤(G)的DNA形成稳定的G-四链体结构,钡离子(Ba2+)也有类似的效应,但与G-四链体形成的络合物稳定性比铅离子络合物差,因此两种络合物穿越纳米孔的阻滞时间不同,基于此即可同时进行两种离子的纳米孔检测[59]。 这种基于DNA碱基与金属离子相互作用的纳米孔分析方法,可拓展到任何与DNA有相互作用的离子和分子的检测研究中。
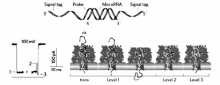
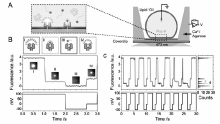
纳米孔分析技术能够在单分子水平通过电流信号提供分子的信息,光谱技术能实时提供分子结构信息,为纳米孔检测提供强有力的信息支持。 目前,单个纳米孔测序的效率不高,为了满足人类基因组尺度的高速纳米孔测序,一个超高通量的纳米孔测序阵列将是极为有用的。 现有的基于膜片钳的电测量手段在技术和成本方面难于进行高通量测量,Huang等[60]开发了具有通量优势的光学纳米孔成像技术,并应用于核酸序列识别(图15)。 该纳米孔阵列具有每平方毫米达到104个纳米孔的检测密度,能够达到等效于pA级别的检测精度,可以对DNA序列和miRNA序列进行定性识别。 同时,该光学纳米孔阵列也可以被做成一个具有2500个磷脂膜的纳米孔检测芯片,实现多种生物样品的高速,高通量,多样品的单分子纳米孔测量。
生物纳米孔分析技术已广泛应用于DNA测序、蛋白质结构研究及单分子检测等领域,且应用领域正在不断拓宽。 然而,作为一种新颖的单分子分析手段,纳米孔技术在实际样品检测、数据处理和检测装置等方面也存在着诸多挑战。
第一,提高生物纳米孔分析技术的选择性与灵敏度。 可以从以下几个角度进行:1)设计不同的高灵敏分子识别探针,以期用于实际复杂样品的检测;2)与质谱联用,结合质谱提供的单分子定性信息,从而实现生物纳米孔单分子质谱;3)将纳米孔电分析技术与光谱技术结合。光谱技术能实时提供分子结构信息,为纳米孔检测提供强有力的信息支持。 纳米孔分析技术结合单分子荧光的工作已有报道,相信在未来的发展过程中,更多的光学技术会进入该领域,以期获得多重单分子行为信息,实现多层次、多角度地对待测物进行实时、动态、在线分析研究。
第二,优化数据处理程序。 生物纳米孔分析技术的数据处理具有数据量大、信号形态复杂、处理手段繁琐、耗时等特点,这些问题阻碍了纳米孔分析技术从科学研究到实际应用的快速转化。 一个理想的数据分析系统应该具备对复杂庞大的实验数据进行快速读取、信号识别、分类、统计并形成图谱的功能。 开发智能的数据分析软件实现精确快速的数据分析是十分必要的。 Long等[61]首次提出“积分还原电流”思想的方法进行电流信号校准,有效还原了受仪器带宽限制的失真纳米孔道单分子信号,显著提高了数据处理的准确度。 进一步,通过引入具有大数据自学习能力的隐马尔科夫算法实现了在较高噪音的数据中纳米孔道电信号的智能聚类识别分析[62],从而开发了一种快速自动化的数据处理软件(http://people.bath.ac.uk/yl505/nanoporeanalysis.html)。 随着数据分析软件的不断进步,生物纳米孔分析技术中的数据分析在简便化的同时也会向更加合理化和标准化的方向迈进。
第三,开发高时间分辨、高电流分辨的纳米孔检测装置。 纳米孔生物传感分析技术所能达到的时间分辨率、电流分辨不但与纳米孔自身的尺寸、实验环境有关,也与检测仪器的性能有直接关系。 在生物纳米孔单分子检测中,我们希望仪器同时具有高电流分辨和高时间分辨。 高电流分辨率可以捕捉电流信号中微小的波动,而高时间分辨率则可保证分析物穿过纳米孔时的所有信息都被完整地记录下来。 目前,被用于纳米孔分析的传统膜片钳系统在仪器技术方面尚无法完全满足纳米孔分析技术的检测要求,因此开发适用于纳米孔分析、满足DNA测序要求的仪器也成为该领域今后发展的重要方向。 另一方面,在传统膜片钳系统中,电流放大器和数模转换器尺寸均很大,很难做到便携式并应用于现场快速检测。 目前,国内外一些课题组已经针对该方面开始了相关研究工作,英国Oxford Nanopore Technologies公司一直致力于发展基于纳米孔的DNA测序仪,其小型化的MinION已经在2014年的全球基因组生物学与技术进展大会(AGBT)上正式亮相。 为实现纳米孔道瞬态电流信号的精准获取,Long等[63]通过集成化设计,开发了具有低噪音高带宽的纳米孔单分子分析系统,从而实现了对生物纳米孔瞬态信号的高时间分辨及高电流分辨。
随着生物纳米孔检测体系的不管完善以及与其它传感技术的交叉融合,相信纳米孔传感未来将大大推动基因测序及个性化医疗的发展。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|
| [47] |
|
| [48] |
|
| [49] |
|
| [50] |
|
| [51] |
|
| [52] |
|
| [53] |
|
| [54] |
|
| [55] |
|
| [56] |
|
| [57] |
|
| [58] |
|
| [59] |
|
| [60] |
|
| [61] |
|
| [62] |
|
| [63] |
|